Des chercheurs de Microsoft ont créé des « étiquettes moléculaires » à partir de brins d’ADN présynthétisés et déshydratés. Un marquage peu coûteux, infalsifiable et pouvant être intégré à n’importe quel support de manière invisible. De quoi remiser aux oubliettes le sacro-saint code-barres ?Vous aimez nos Actualités ?Inscrivez-vous à la lettre d’information La quotidienne pour recevoir nos toutes dernières Actualités une fois par jour.Cela vous intéressera aussi

les secrets de l’ADN L’ADN est porteuse de l’information génétique. Chaque cellule possède un noyau constitué de chromosomes contenant de l’ADN. Nous avons interviewé Jean-Louis Serre, professeur de génétique, pour qu’il nous parle plus en détail de cette molécule contenue dans tout organisme vivant.
Une série de barres noires et blanches agrémentée d’un numéro à 10 chiffres : le code-barres, inventé dans les années 1970, a révolutionné le commerce. Les années 2000 ont vu l’avènement du QR code (pour Quick Response code, en anglais), inspiré par le design du jeu de Go. Les années 2020 pourraient être celles du code « porc-épic », tel que l’ont surnommé les chercheurs de l’université de Washington et de Microsoft Research. Leur idée, décrite dans Nature Communications, n’est pas entièrement nouvelle : il s’agit de coder des informations sous forme d’ADN, où les bits informatiques 0 et 1 sont remplacés par les nucléotides A, C, G et T. Cette technique permet théoriquement de stocker beaucoup plus d’informations sur un espace réduit et de les conserver extrêmement longtemps (pendant plusieurs millions d’années).
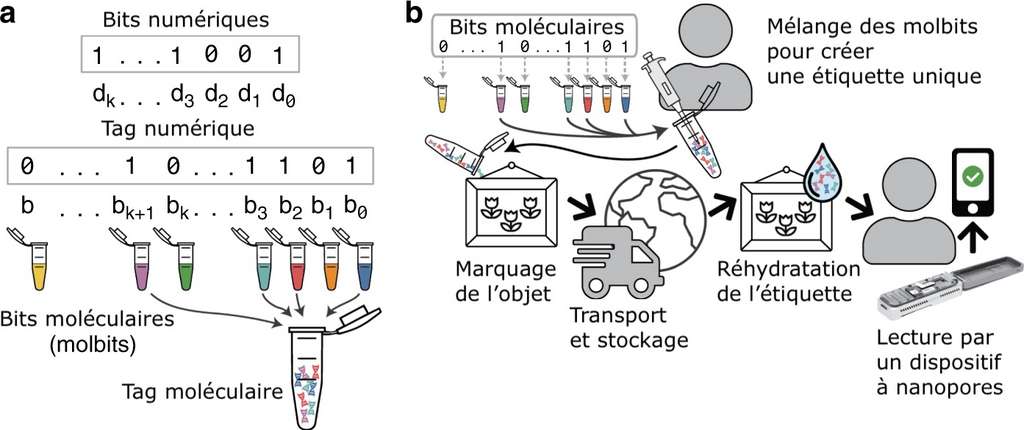
Le système Porcupine expliqué. Les bits informatiques sont convertis en « molbits », des bits moléculaires, sur des brins d’ADN présynthétisés. Ces molbits sont mélangés pour créer une étiquette unique sous forme d’ADN déshydraté, qui peut être apposée sur un objet. Une fois réhydratée, la séquence ADN est lue en quelques secondes via un appareil portable à nanopores. © d’après Kathryn Doroschak et al, Nature Communications, 2020
Des « molbits » moléculaires pour remplacer les bits informatiques
Le problème, c’est que le cryptage et le décryptage de l’ADN sont longs et coûteux. « Les méthodes existantes sont encore compliquées et nécessitent l’accès à un laboratoire, ce qui exclut de nombreuses applications dans le monde réel », témoigne Kathryn Doroschak, bio-informaticienne à l’université de Washington et auteure principale de l’étude. Pour simplifier le process, le système « Porcupine » (porc-épic, en français) se base sur des fragments d’ADN prédéfinis encodés sur 96 « molbits », des bits moléculaires. Ces molbits peuvent ensuite être mélangés arbitrairement pour créer de nouveaux marqueurs moléculaires. « À partir des 96 codes-barres initiaux, Porcupine peut produire environ 4,2 milliards d’étiquettes uniques sans compromettre la fiabilité de la lecture », détaille Karin Strauss, coauteure de l’étude et chercheuse chez Microsoft Research. Ces brins présynthétisés permettent de réduire notoirement les coûts, ce qui rend le système accessible pour des applications grand public. Un dispositif portable à nanopores est ensuite utilisé pour programmer et décoder ces étiquettes en quelques secondes.VOIR AUSSIMicrosoft : une machine qui transforme les bits informatiques en ADN
Des étiquettes invisibles et infalsifiables
Un tel marquage ADN présente de nombreux avantages. La déshydratation des brins après l’assemblage initial de l’étiquette permet de prolonger la durée de vie de l’étiquette et d’éviter la contamination par d’autres ADN présents dans l’environnement. De plus, comme les molbits ne mesurent que quelques centaines de nanomètres de long, « un milliard d’étiquettes peuvent tenir dans un seul millimètre carré », rapporte Jeff Nivala, de l’université de Washington. On peut ainsi les intégrer sur des surfaces minuscules ou flexibles, sur lesquelles il serait impossible de coller une étiquette (par exemple intégrer une étiquette de prix directement dans un vêtement). Comme elles sont invisibles, ces étiquettes « porc-épic » ne peuvent être détectées ni trafiquées. « Elles sont donc idéales pour suivre des articles de valeur et protéger les marchandises contre les contrefaçons, poursuit Jeff Nivala. On pourrait aussi utiliser un tel marquage moléculaire pour suivre les bulletins de vote des électeurs et empêcher toute falsification lors de futures élections. » De quoi empêcher Donald Trump de crier à la fraude ?
De l’ADN en code-barres
ne nouvelle technique d’identification génétique a été développée dans des laboratoires suédois et danois. Notre génome pourrait donc être cartographié par nanofluidique sous la forme de 46 codes-barres, autant que de chromosomes.
L’ADN est un outil précieux dans plusieurs domaines : la médecine, pour le dépistage de maladies génétiques ou d’agents infectieux, mais aussi en justice puisqu’il peut permettre de confondre irréfutablement les auteurs de crimes.
Depuis la découverte de l’ADN, les méthodes d’identification des séquences sont passées de l’artisanat, avec le séquençage de seulement quelques centaines de nucléotides par jour (méthode de Maxam-Gilbert), à des méthodes industrielles automatiques où les machines peuvent traiter 600 millions de paires de bases (pyroséquençage 454).
Une nouvelle technologie vient de naître, rapide et peu coûteuse, qui pourrait permettre d’accélérer l’obtention d’informations génétiques. De plus, la technique est précise, puisqu’elle s’attaque à une molécule unique d’ADN, et non plus à un ensemble de molécules comme c’est actuellement le cas.
La technique, développée par nanofluidique à l’Université de Lund en Suède, permet d’obtenir une image de l’ADN sous forme de code-barres, spécifique de la composition nucléotidique de l’ADN. Elle débute par le dépliage de chacun des chromosomes contenus dans une cellule, dans un nanotunnel dessiné sur une puce. Ensuite, elle repose sur les propriétés physiques de l’ADN.
Les quatre nucléotides (les bases de l’ADN : l’adénine, la thymine, la cytosine et la guanine), forment des paires de bases qui relient les deux brins de l’ADN. L’adénine s’apparie à la thymine dans une liaison plus labile que celle formée par la cytosine et la guanine. Ces différences mènent à une cassure des liaisons A-T à une température plus basse que les liaisons G-C. En fonction de la température appliquée à la molécule d’ADN, les deux brins de l’ADN sont alors plus ou moins appariés, à l’image d’une fermeture à glissière cassée, qui est ouverte ou fermée en différents endroits.
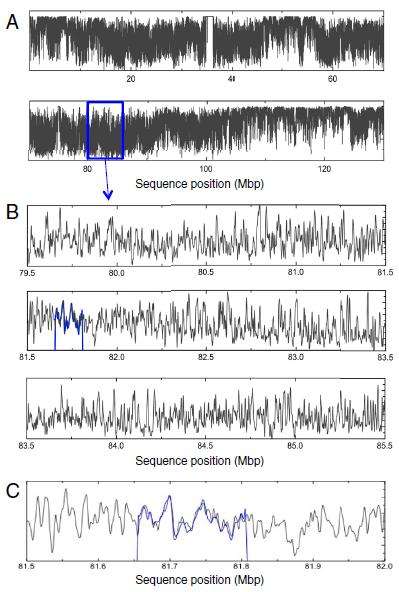
Visualisation, à plus ou moins grande échelle (agrandissement de A à C), des différences d’intensité de fluorescence en fonction du positionnement sur le chromosome 12 humain. A. Profile de fusion du chromosome entier. B. Profile de fusion de la position 79.500.000 à la position 85.500.000. C. Agrandissement du profile de fusion de 500 kilopaires de bases. La courbe bleue superposée correspond à une partie de chromosome 12 clonée. © Université de Lund / PNAS
De nombreuses applications possibles
Pour distinguer les zones appariées des zones ouvertes, une molécule fluorescente particulière a été utilisée : elle est séquestrée dans les zones doubles-brins de l’ADN. La fluorescence est donc plus forte aux endroits où les deux brins sont appariés. Ce sont les différences de fluorescence sur toute la longueur de la molécule d’ADN qui dessinent le code-barres.
La technique, publiée dans le journal PNAS, semble très au point, puisque les chercheurs ont comparé les codes-barres obtenus expérimentalement à ceux calculés de manière théorique : la superposition est quasi-parfaite. Toutefois, seule une image globale de l’ADN est obtenue : il n’est pas possible de déterminer la séquence nucléotidique exacte.
Ses applications potentielles sont néanmoins nombreuses : des bases de données pourraient contenir tous les ADN connus sous la forme de code-barres, afin de faciliter l’identification de virus ou de bactéries présents chez un patient. Elle pourrait aussi aider à déterminer des anomalies chromosomiques, souvent causes de maladies génétiques, en comparant le code-barres d’un chromosome sain à celui d’un patient.
La technique, qui permet de visualiser individuellement chaque chromosome d’une seule cellule, peut aussi déterminer des différences génétiques entre deux cellules au sein même d’une population cellulaire, chose irréalisable avec les autres technologies de séquençage. C’est par exemple important pour comprendre la résistance au traitement de cellules cancéreuses, dont l’hétérogénéité génétique pourrait être en cause.
 Rewmi.com L'Equilibre notre Crédo
Rewmi.com L'Equilibre notre Crédo